Desde que la experimenté por primera vez, siempre me ha parecido que hay algo realmente mágico en la secuenciación de ADN. Como sistemático, se supone que estás familiarizado con el organismo del que extraes tu muestra, lo conoces, a veces hasta en sus más íntimos detalles. En un momento dado es un pellizco de tejidos en un tubo de plástico, y unos minutos después desaparece de tu vista y se queda en unos ridículos 100 microlitros (apenas un goterón) de un líquido aparentemente indistinguible del agua. No voy a decir que es fe lo que mantienes durante todo el proceso de secuenciación (daría lugar a malentendidos), pero con una confianza «a ciegas» en el método, consigues una secuencia de As, Ts, Gs y Cs después de que las enzimas y los sabios cambios de temperaturas hayan obrado su milagro. Días o semanas después, frente a un ordenador, llega la prueba del algodón. La secuencia de nucleótidos por sí no te dice nada, pero alineada en su lugar correspondiente, hace que todo cobre sentido: surge un patrón o una relación (esperada o no), que no es producto de la casualidad, sino de la evolución, de la relación de ancestros comunes que nos tiene a todos unidos. Nadie había obtenido antes esa secuencia de ese individuo, y pese a todo la homología esperable con sus secuencias hermanas es indiscutible; se te revela de forma privada un secreto secular. Mágico, de verdad.
Estos días, toda mi relación con la secuenciación de ADN está sufriendo un cambio espectacular gracias a la memorable toma de contacto que estoy teniendo con la llamada «secuenciación de nueva generación». Cuando termine el «entrenamiento» podré utilizar este encantador cacharrito:
Hoy me gustaría contaros por qué estoy enamorado de esta suerte de panificadora gigante, y por ello quiero explicaros la abismal diferencia, cualitativa y cuantitativa que suponen los nuevos métodos de secuenciación respecto a lo que se había venido haciendo hasta entonces.
Empecemos por el principio:
Amplificar y secuenciar ADN es técnicamente muy sencillo, basta seguir las recetas al uso. Sin embargo lo apasionante es entender cómo y porqué tiene lugar el milagro. Durante varias décadas, los laboratorios de todo el mundo han venido usando técnicas muy parecidas para realizar este proceso. No entraré en mucho detalle, pues los que estéis familiarizados con ellas las conoceis de sobra y para los que no bastarán algunas pinceladas.
Secuenciación como la conocíamos (hasta hace unos años)
Entre tener delante una muestra y leer una secuencia de su genoma normalmente hay tres pasos: extracción, amplificación y secuenciación.
Extracción: Muy asequible gracias a protocolos sencillos en los que se machaca un tejido, se le somete a la acción de un detergente (que digiere las estructuras) y luego se filtra el puré resultante reteniendo el ADN en membranas especiales, resuspendiéndolo al final. Con esto se obtienen extractos de todo el genoma ahí apelotonado.
Amplificación: Normalmente no es posible secuenciar todo el genoma, sino que se elige una región concreta que nos interese y tratamos de obtener una suspensión muy pura que contenga sólo esa región. Esta amplificación se hace normalmente por Reacción en Cadena de la Polimerasa (PCR en inglés). Se toma una pequeña alícuota del extracto de ADN, se le añaden unos fragmentos pequeños (cebadores) que delimitan la región que nos interesa, unas enzimas, y cambiando sabia y cíclicamente la temperatura se consigue que dichas enzimas repliquen exponencialmente la región a secuenciar.
Típicos tubos de PCR, para que os hagais cargo del volumen empleado en cada reacción, del que puede resultar una única secuencia de unos pocos cientos de pares de bases
Secuenciación: El producto de la PCR, una vez purificado, se somete una vez más a perrerías enzimáticas que consiguen hacer muchas copias del fragmento, pero de distinta longitud y con el nucleótido terminal «coloreado» de manera diferencial según sea A, T, C o G. Mediante una electroforesis en geles capilares se detectan los fragmentos ordenados por tamaño, y leyendo el «color» del nucleótido terminal, se reconstruye finalmente la secuencia. Este sistema lleva siendo la quintaesencia de la secuenciación desde hace 30 años y se conoce como «método Sanger«.
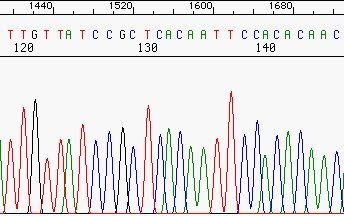
Típico cromatograma Sanger, de toda la vida
Hagamos cuentas: cada tubito de reacción de PCR (normalmente con un volumen de reacción de 25 microlitros) permite conseguir una única secuencia de pocos cientos de pares de bases de longitud. Además, generalmente las secuencias deben ser revisadas y editadas manualmente (aunque es cierto que ya hay programas informáticos estupendos que reducen muchísimo el trabajo, virtualmente el investigador revisaba base a base la secuencia, si era necesario). Tanto la PCR como el método Sanger son muy ingeniosos y es muy interesante conocerlos a fondo tanto en su funcionamiento como en los detalles de su desarrollo por Mullis y Sanger respectivamente. Si no lo conocéis os animo a que profundicéis a partir de los enlaces a la Wikipedia.
En definitiva: dependiendo del equipamiento del laboratorio, la secuenciación a la antigua usanza permitía, si se hacía a tumba abierta, ir sacando del orden de miles o como mucho decenas de miles de pares de bases a la semana, que exigían luego cierto trabajo de edición manual. Los factores limitantes se hallaban tanto en el trabajo manual de preparación de «volúmenes de amplificación» (tan sólo una reacción por tubo, que debe ser preparado manualmente) así como en la capacidad de los secuenciadores Sanger (normalmente capaces de secuenciar en tandas de bandejas de 96 pocillos) y por supuesto, en el tratamiento bioinformático de las secuencias. Ninguna de estas cuestiones era moco de pavo.
Secuenciación por síntesis
En los últimos cinco años se han venido multiplicando una serie de métodos que han pulverizado sistemáticamente las mejores perspectivas para obtener información genómica. Aquí muestro sólo un ejemplo, el de la técnica con la que me estoy familiarizando, pero hay más. Paso de decir el nombre del cacharro, que bastante publicidad le estoy haciendo ya, cosa que no le hace falta a la compañía de turno que ya de por sí se comporta como el «Apple» del mundo de la secuenciación, aunque curiosamente su software corra en Windows y/o Linux (no le pega nada).
Todo lo que aprendo de este chisme me deja con la boca abierta. Es ACOJONANTE. Todo está asombrosamente automatizado. Básicamente, haces un tratamiento previo a tu muestra (unas pocas horas), la cargas una sola vez en un cartucho de reactivos, lo metes en el cacharrín, le das a un botón y te vas a tomar unas birras. En cuestión de horas, el bicharraco te secuencia del orden de miles de millones de pares de bases, a partir de una única muestra y te las ofrece alineadas, sin necesidad de edición manual y con una precisión que deja con la boca abierta. De una sola tacada se pueden secuenciar genomas pequeños completos (por ejemplo, bacterianos o mitocondriales).
La química del proceso se explica sucintamente en este vídeo.
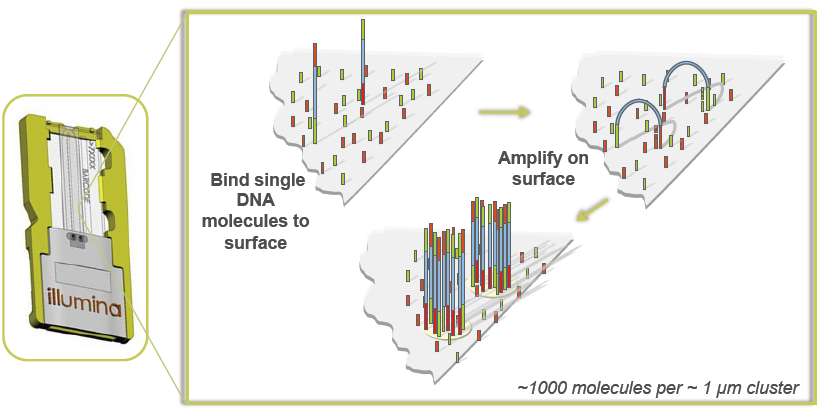
Resumiendo: la muestra pretratada (o biblioteca de ADN) se carga sobre un canal en una diminuta placa transparente por la que se puede controlar con gran precisión el flujo de distintos reactivos y cambios de temperatura. La superficie está tapizada de oligonucleótidos a los que se unen de forma aleatoria los fragmentos de la biblioteca gracias a los adaptadores que se les ha insertado durante su preparación. El dispositivo está montado de tal manera que se pueden realizar amplificaciones in situ de los fragmentos de ADN de una forma similar a una PCR ordinaria, pero en lugar de quedar en suspensión, cada fragmento se amplifica localizadamente sobre la superficie física de la célula de flujo, originándose un campo de «pincelillos», cada uno de ellos siendo un conjunto de tan sólo unas 1000 hebras de ADN idénticas. El problema del control de los volúmenes queda resuelto de esta forma: hasta 800.000 de estos pincelillos (clusters) en un milímetro cuadrado.
Célula de flujo del cacharrito en cuestión. Tamaño similar al de un sello. Tremendo
¡Los pincelitos!
La fase de secuenciación se resuelve por un sistema denominado «secuenciación por síntesis». Las hebras de ADN se van completando con nucleótidos fluorescentes, con la particularidad de que están «capados» (no se puede continuar la síntesis después de ellos), pero de forma reversible. De esta manera, y una vez más gracias a un control absoluto de los tiempos y de los flujos de los distintos reactivos por la célula, la síntesis se hace nucleótido a nucleótido, simultáneamente, y emitiendo la luz que corresponde.
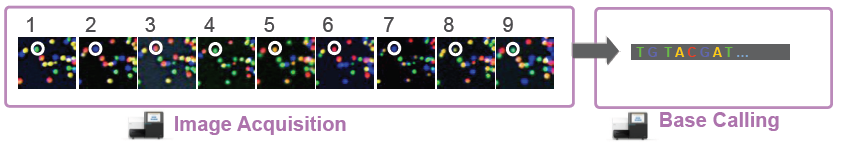
El equipo óptico se encarga de tomar instantáneas en cada uno de los ciclos, reconociendo el nucleótido que se ha añadido, e integrándolo en su respectivo archivo. La precisión de este sistema es espectacular, mucho mayor que con el método Sanger, y hace innecesaria una edición manual de la secuencia… ¡y menos mal! ¡Porque con el volumen de datos producidos sería poco menos que imposible! Al igual que pasa con otros métodos de secuenciación a lo bestia, originalmente no hay conocimiento sobre qué región del genoma representa cada pincelillo, sino que es el alineamiento y ensamblaje posteriores de millones y millones de fragmentos el que acaba originando porciones reconocibles al comparar con otros genomas de referencia.
Esta es una imagen de uno de los destellos de un nucleótido determinado en una loseta. Cada punto es la imagen producida por uno de los pincelillos
De verdad que da vértigo comprobar a lo que hemos llegado. Es impresionante el alarde de nanotecnología de óptica y fluidos, no tengo palabras. El chisme en cuestión creo que viene a costar unos 125.000 dólares, que puede parecernos caro, pero no lo es tanto. Incluso el coste de hacer un análisis en él (preparación de la biblioteca incluida) es bastante razonable si se tiene en cuenta lo baratísimo que sale el precio por base en comparación con el método Sanger.
Lo preocupante quizá sea otra cuestión. La primera vez que supe sobre los nuevos métodos de secuenciación fue en 2008, e igualmente me quedé impresionado por el funcionamiento de un método distinto: la pirosecuenciación, implementada por una compañía distinta y usando otros fundamentos. Bien, pues aunque nadie lo dice, todo parece indicar que la pirosecuenciación se está quedando obsoleta. Es tremendo pensar que la PCR y el método Sanger de toda la vida se han venido usando durante décadas, y sin embargo ahora da la impresión que un novísimo sistema puede tener una vida media mucho más corta. No es que me parezca mal, pero seguro que a los laboratorios que se compraron el chisme de turno no les hace tanta gracia.



Corta corta…En un curso de verano un señor que hacia negocio con estos cacharros nos habló del «Oxford Nanopore» Una especie de illumina más rápido y en miniatura que según nos decía «va a llegar pronto» Asombroso tan lejano e irónicamente tan «cercano».
Saludos !!
Muy buena entrada compañero y menuda envidia me das macho. Yo también quiero la termomix esa que te ILUMINA mientras te vas de cañas jeje.
A mi también me da la sensación de que cada vez las técnicas se pasan de moda más rápido. Y lo he dicho bien, se pasan de moda. No es que no sean útiles, porque Sanger se sigue usando a diario y es de mucha utilidad y barato para el uso cotidianos de construcción de mutantes. Pero se pasan de moda y los correctores de artículos te piden la última técnica que está mas en boga.
En fin, lo dicho, me das envidia sana y me ha gustado mucho la entrada.
Todos los que rondamos «el mundillo» nos apasionan estos cacharros. Más cuando se han realizado secuenciaciones completamente a mano con esos geles de poliacrilamida en los cristalazos…ainsss…¡qué tiempos! :D
En fin, lo que es alucinante es la bajada de precios. Recuerdo que hace 3 años mostré en mi blog como el BGI (Instituto de Genómica de Pekín) adquirió 128 Illumina Seq2000. Y de aquellas costaban unos 650.000 dólares.
Enhorabuena por el artículo y más aún por tu «training». Un abrazo ;)
Raven: «Oxford Nanopore», guau, le he echado un ojo a la página web y sí, parece prometedor. Nadie se salva, ahora parece que los de Illumina están que lo petan, pero no parece haber límite, es impresionante.
Scarios HR: totalmente de acuerdo, lo mismo me he pasado un poco y ahora parece que nadie usa secuenciación Sanger. Para muchas cosas sigue siendo lo mejor, no se trata de matar moscas a cañonazos. Quería además animar a quien no lo conozca a que se entere de cómo funciona el proceso, que es realmente ingenioso.
Dr.Genoma: ¿Has leído secuencias en geles de acrilamida? Guau. Oye, he visto el post que dijiste (algo se cuece en China, jajaja) y me ha gustado lo del mapita que enlazabas.
Pingback: La especie de la semana: Rhacophorus helenae, con comentario crítico sobre las especies crípticas « Diario de un copépodo
Mágico, dices? Mágico! Supercherías! Herejía! Y no me vengas con destellos de nucleótidos y la mar en coche, esas fotos son de una tele de las viejas con lluvia por falta de programación.
¡¡¡Chick va a enterarse de esto!!! (atento a los tres signos de exclamación)
Flipo. He trabajado con secuenciadores clásicos, los de capilar, y allá en la carrera incluso con los primeros métodos, radiactividad, geles de acrilamida, etc… Cacharritos de estos hacen que me quede anonadado.
Y pienso lo mismo que tú, ¿qué nos espera mañana? Porque sí, te compras el aparato de 125.000 dólares y en dos años es muy probable que esté obsoleto o casi.
Je, la ciencia avanza que es una barbaridad :-) Salud y saludos.
Jopelines, cómo avanzan los tiempos, qué barbaridad… en mi época con cien pesetas… uy, que se me va el discurso, aunque no tanto.
Mañana tengo que llevar unas muestras a secuenciar, y por tu culpa casi que me da vergüenza. Pero bueno, en realidad el punto de sorpresa también vale para lo que comentan los parroquianos, a mi particularmente me alucina lo poco que vale «de dinero» y lo poco que cuesta «de tiempo» hoy día, hacer una purificación cutrecilla de ADN, subirlo al piso de arriba y que al día siguiente tengas (si todo va bien) una secuencia la mar de maja para comprobar que tu secuencia está como toca. Cuando antaño, secuenciar un gen ¡podía ser una tesis entera!.
En fin, que disfrutes del cacharrito y gracias por compartirlo: me ha emocionado el primer párrafo del post, y no es coña; me gusta mucho pararme a pensar en lo «mágico» y fascinante de las cosas que hacemos cotidianamente en el laboratorio.
Muy interesante.
Yo de esto no sabia nada. Lo unico que me sonaba parecido es que estaba intentando estudiar enzimas de extremofilos para hacer la PCR en caliente y por lo tanto más rapida.
No te sientes un poco como en parque jurasico?
Quien se acuerda cuando se preveia que el proyecto Genoma humano, se iba finalizar en el 2015? De repentes unas cuantas empresas privadas se pusieron a competir y a secuenciar a unas velocidades inimaginables.
No sé si conocias el metodo casero para extraer ADN de una Cebolla con zumo de piña?
La nanofontaneria de esa maquina me ha recordado a las computadoras de burbujitass.
Pancho: chico, mágico mágico… no te lo tomes tan a pecho, es que estoy todavía en la fase de encoñamiento. Siento nucleótidos en el estómago.
Exseminarista, Dr. Litos: gracias por comentar, majos. Si ya sé yo que muchos de vosotros me entendéis perfectamente.
Pipistrellum:: gracias por el vídeo, pero sobre todo, gracias por el enlace de la cebolla. Lo conocía de oídas, pero así está mucho mejor explicado.
Ya no te mando, mas links son los ultimos sobre el tema.
http://naukas.com/2012/09/24/los-transistores-fluidicos/
http://en.wikipedia.org/wiki/Nanofluidic_Circuitry
Pingback: Los osos polares que se volvieron pardos a base de polvos | Diario de un copépodo